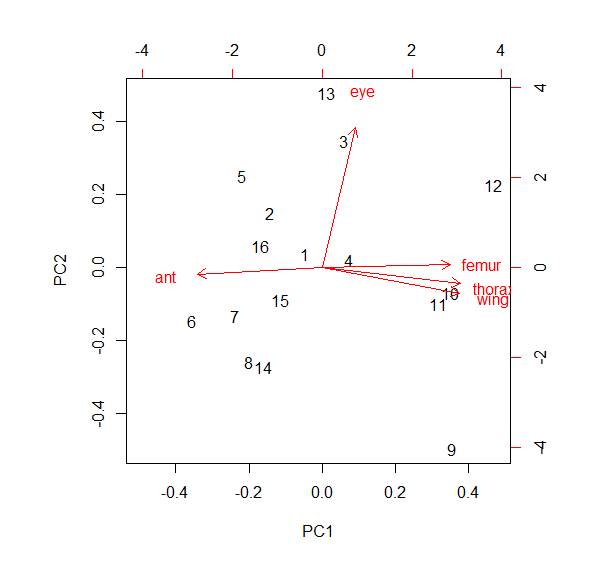
#資料有大於兩種互相獨立的分組方法
#多因子檢定 (Multifatorial testing): 需分析因子數越多,需要分析的交互作用項也會快速增加。
#三因子變異數分析 (無重複試驗) [Three-way ANOVA (without replication)]
#Exzample: 白羅曼鵝的性別、受日照長短、試驗區域對其採食量影響
intake<- c(78.1,76.3,
69.5,73.2,
82.4,83.0,
72.3,70.0)
sex<- rep(c("F","F","M","M"),times= 2)
light<- c("L","L","L","L","S","S","S","S")
region<-c("A","B","A","B",
"A","B","A","B")
data.frame(intake=intake, sex=sex, light=light,region=region)
summary(aov(intake~sex*light*region))
summary(aov(intake~sex*light*region-sex:light:region)) #為無重複試驗時須要扣除同時有三因子之交互作用 (-sex:light:region)